Utilisation de l'API Python-netcdf4
Générer des fichiers NetCDF avec Python
netCDF est un format "binaire" qu'on ne peut donc pas éditer ou créer avec des outils de bureautique classiques. Il faut donc maîtriser un ou plusieurs outils qui permettent de créer des fichiers netCDF.
La creation basique d'un fichier netCDF peut se faire comme on l'a vu précédemment avec les outils de base "ncgen" à partir d'un fichier texte "cdl". mais cette solution peut etre convenable pour modifier un fichier, pas pour le créer "ex nihilo"
- La solution de création par programmation s'impose assez rapidement quand on des calculs et des transformations à faire depuis des fichiers de capteurs par exemple, ou des fichiers de données.
Pour cela netCDF fournit une API (interface de programmation) qui est utilisable depuis de nombreux langages comme Python, R, java, C, C++, Fortran, Perl, Ruby, etc...
voir:
- https://docs.unidata.ucar.edu/netcdf-c/current/netcdf_apis.html
- https://www.unidata.ucar.edu/software/netcdf/software.html
Nous donnons ci dessous un exemple d'utilisation de l'API python-netcdf dont les principes peuvent etre exportés
dans d'autres langages.
Installation de la librairie python3-netCDF4
Documentation de l'API Netcdf:
- la Documentation de l'API Netcdf se trouve ici : https://unidata.github.io/netcdf4-python/
# sur Linux Debian, Ubuntu, Mint : avec le paquet de la distribution linux
sudo apt-get install python3-netcdf4
# ou directement le paquet python
pip install netCDF4.
# sur MacOS
- https://formulae.brew.sh/formula/netcdf
- brew install netcdf4
# sur Windows
https://downloads.unidata.ucar.edu/netcdf-c/4.9.2/netCDF4.9.2-NC4-DAP-32.exe
https://docs.unidata.ucar.edu/netcdf-c/current/winbin.html
Rappel sur les sections d'un fichier netCDF
- une section "dimensions"
- une section "variables"
- une section "global attributes"
- une section "data"
- la section "dimensions" et "variables" du header
netcdf bathybot_MICROCAT_20240128 {
dimensions:
time = UNLIMITED ; // (480 currently)
lenstation = 56 ;
variables:
char station_id(lenstation) ;
station_id:standard_name = "platform_name" ;
station_id:long_name = "station name" ;
station_id:technical_data = "true" ;
station_id:comment = "internal name of the station" ;
station_id:units = "Not applicable" ;
station_id:coordinates = "TIME; DEPTH; LATITUDE; LONGITUDE" ;
station_id:sdn_parameter_name = "Subsample identifier" ;
station_id:sdn_parameter_urn = "SDN:P01::SSAMID01" ;
station_id:cf_role = "timeseries_id" ;
float LATITUDE ;
LATITUDE:units = "Degrees north" ;
LATITUDE:comment = " " ;
LATITUDE:coordinates = "TIME; DEPTH; LATITUDE; LONGITUDE" ;
LATITUDE:standard_name = "latitude" ;
LATITUDE:axis = "Y" ;
LATITUDE:coverage_content_type = "coordinate" ;
LATITUDE:long_name = "Latitude of measurements" ;
float TEMP(time) ;
TEMP:long_name = "temperature" ;
TEMP:standard_name = "sea_water_temperature" ;
TEMP:units = "Degrees Celsius" ;
TEMP:coordinates = "TIME; DEPTH; LATITUDE; LONGITUDE" ;
TEMP:ancillary_variables = "TEMP_QC" ;
TEMP:FillValue = -999.99 ;
TEMP:sdn_parameter_name = "Temperature of the water body" ;
TEMP:sdn_parameter_urn = "SDN:P01::TEMPPR01" ;
TEMP:sdn_uom_name = "degree_Celsius" ;
TEMP:sdn_uom_urn = "SDN:P06::UPAA" ;
TEMP:sensor_model = "Sea-Bird SBE 37 MicroCat SMP-CTP (submersib
le) CTD sensor" ;
TEMP:sensor_reference = "http://vocab.nerc.ac.uk/collection/L22/
current/TOOL1457" ;
TEMP:sensor_SeaVoX_L22_code = "SDN:L22::TOOL1457" ;
TEMP:sensor_manufacturer = "Sea-Bird Scientific" ;
TEMP:sensor_manufacturer_uri = "http://vocab.nerc.ac.uk/collecti
on/L35/current/MAN0013/" ;
TEMP:sensor_manufacturer_urn = "SDN:L35::MAN0013" ;
- La section des "attributs globaux" dans le header
// global attributes:
:date_created = "29/01/24 07:25" ;
:title = "EMSO Western Ligurian : Bathybot, Microcat sensor (Net
CDF files from 2023-04-28 )" ;
:Conventions = "OceanSITES v1.4,SeaDataNet_1.0,COARDS, CF-1.6" ;
:institution_edmo_code = "3078" ;
:institution_edmo_uri = "https://edmo.seadatanet.org/report/3078
" ;
:institution_ror_uri = "https://ror.org/05258q350" ;
:geospatial_lat_max = "42.8015" ;
:geospatial_lat_min = "42.8014" ;
:geospatial_lon_max = "5.9828" ;
:geospatial_lon_min = "5.9826" ;
:geospatial_vertical_max = "2420" ;
:geospatial_vertical_min = "2420" ;
:time_coverage_start = "2023-04-28T00:00:00Z" ;
:time_coverage_end = "now" ;
:update_interval = "daily 24 hours" ;
:site_code = "Bathybot" ;
:emso_facility = "Western Ligurian Sea" ;
:source = "sea bed vehicle" ;
:platform_code = "\"\"" ;
:wmo_platform_code = "unknown" ;
:format_version = "1.4" ;
:network = "EMSO-ERIC" ;
- la section "donnees"
data:
station_id = "EMSO_Western_Ligurian_Bathybot : Lat 42.8014 Long 5.9826" ;
LATITUDE = 42.8014 ;
LONGITUDE = 5.9826 ;
POSITION_QC = 1 ;
DEPH = 2420 ;
DEPH_QC = 1 ;
TIME = 1706400049, 1706400229, 1706400409, 1706400589, 1706400769,
1706400949, 1706401129, 1706401309, 1706401489, 1706401669, 1706401849,
1706402029, 1706402209, 1706402389, 1706402569, 1706402749, 1706402929,
 On va retrouver dans l'API les différentes fonctions nécessaires pour traiter chacune de ces sections netCDF
On va retrouver dans l'API les différentes fonctions nécessaires pour traiter chacune de ces sections netCDF
- On peut découper la création d'un fichier netCDF en 5 phases principales qui vont correspondre aux différentes sections du fichier, et à des fonctions du programme.
- 1/ Créer le dataset netCDF vierge
- 2/ Créer les dimensions des variables (vecteurs 1D, matrice 2D)
- 3/ Créer les variables dimensionnées
- 4/ Ecrire les métadonnées dans l’entete netcdf
- 5/ Ecrire les données dans les variables
1. Création du dataset netCDF
Il faut commencer par Importer la librairie netCDF4 dans le programme python
import netCDF4 as nc4
1/ Créer le dataset netcdf “vierge”
outNetCDF=”/chemin/vers/lerepertoireoutNetCDF”
FileName=outNetCDF+”/”+nomfic+".nc"
fileNC=nc4.Dataset(FileName,"w", format="NETCDF4", encoding='latin-1')
avec divers modes d’accès au fichier créé
- “w” : écriture nouveau fichier
- “r” : lecture
- “r+” ou “a” pour “append”
2. Créer les "dimensions" (1D vecteurs, 2D matrices, ou plus)
netCDF4.Dataset.createDimension:https://unidata.github.io/netcdf4-python/#netCDF4.Dataset.createDimension
2/ Créer les dimensions (vecteurs 1D ou matrices 2D)
- Cas d’une série temporelle :
featureType=”TimeSeries”:- Point fixe, mesures en fonction du temps
station_name=”julio”
chlength=len(station_name)
fileNC.createDimension('lenstation',chlength) # longueur chaine de caractere station
fileNC.createDimension('time',size=None) # unlimited pour les mesures de temps
- Cas d’un profil vertical :
featureType = “profile”- Point fixe, temps fixe mesures le long de la profondeur
station_name=”romarin”
lenstation=len(station_name)
# longueur chaine de caractere station
fileNC.createDimension('lenstation',lenstation)
# None=unlimited pour recevoir les mesures sur le profil vertical
fileNC.createDimension('depth',size=None)

Note
Comme on le voit dans les exemples ci dessus, dans l'API netcdf, les chaine de caracteres sont traitées comme des tableaux de valeurs, pour lesquels il faut déclarer une "dimension" qui refletent la longueur de la chaine de caracteres, et qui n'a rien à voir avec les "dimensions" selon lesquelles evoluent les variables mesurées.
3. Créer les variables
netCDF4.Dataset.createVariable : https://unidata.github.io/netcdf4-python/#netCDF4.Dataset.createVariable
- exemple variable "stationname" et "latitude"
# on cree la variable netcdf
stationname = fileNC.createVariable('stationname', 'S1', ('lenstation',))
# et on instancie les différents attributs de la variable
stationname.standard_name = 'platform_name'
stationname.long_name = 'station name'
stationname.cf_role = 'timeseries_id' #obligatoire convention CF
stationname.sdn_parameter_name = "Subsample identifier"
stationname.sdn_parameter_urn = "SDN:P01::SSAMID01"
# on cree la variable netcdf
lat = fileNC.createVariable('LATITUDE','f4' , fill_value=None)
# et on instancie les différents attributs de la variable
lat.units = 'Degrees north'
lat.comment = " "
lat.coordinates = "TIME; DEPTH; LATITUDE; LONGITUDE"
lat.standard_name = 'latitude'
lat.axis = "Y"
lat.coverage_content_type = "coordinate"
lat.long_name = "Latitude of measurements"
lat.sdn_parameter_name = "Latitude north"
lat.sdn_parameter_urn = "SDN:P01::ALATZZ01"
lat.sdn_uom_name = "Degrees north"
lat.sdn_uom_urn = "SDN:P06::DEGN"
lat.ancillary_variables = "POSITION_QC"
- cas de la variable "time"
NC_TIME_FMT = 'seconds since 1970-01-01T00:00:00Z'
# on cree la variable netcdf
timenc = fileNC.createVariable('TIME', 'i4',('time',))
# et on instancie les différents attributs de la variable
timenc.long_name = "time of measurements"
timenc.standard_name = "time"
timenc.axis = "T"
timenc.coverage_content_type = "coordinate" ;
timenc.units = NC_TIME_FMT
timenc.origin = "01-JAN-1970 00:00:00"
timenc.calendar = 'standard'
timenc.sdn_parameter_name = "Elapsed time relative to 1970-01-01T00:00:00Z";
timenc.sdn_parameter_urn = "SDN:P01::ELTMEP01"; #code depuis 1970 sinon c'est ELTJLD01 pour 1950
timenc.sdn_parameter_uri = "https://vocab.nerc.ac.uk/collection/P06/current/UTBB/";
timenc.sdn_uom_name = "seconds";
timenc.sdn_uom_urn = "SDN:P06::UTBB";
timenc.ancillary_variables = "TIME_QC"
timenc.coordinates = "TIME; DEPTH; LATITUDE; LONGITUDE"
- Variable Temperature
# on cree la variable netcdf
TEMP_nc = fileNC.createVariable('TEMP', 'f4',('time'))
# et on instancie les différents attributs de la variable
TEMP_nc.long_name = "temperature"
TEMP_nc.standard_name = "sea_water_temperature"
TEMP_nc.units = "Degrees Celsius"
TEMP_nc.comment=""
TEMP_nc.coordinates = "TIME; DEPTH; LATITUDE; LONGITUDE"
TEMP_nc.ancillary_variables="TEMP_QC"
TEMP_nc.FillValue= -999.99
TEMP_nc.sdn_parameter_name="Temperature of the water body"
TEMP_nc.sdn_parameter_urn="SDN:P01::TEMPPR01"
TEMP_nc.sdn_uom_name="degree_Celsius"
TEMP_nc.sdn_uom_urn="SDN:P06::UPAA"
TEMP_nc.sensor_model=sensor_model
TEMP_nc.sensor_reference=sensor_reference
TEMP_nc.sensor_SeaVoX_L22_code=sensor_SeaVoX_L22_code
TEMP_nc.sensor_manufacturer=sensor_manufacturer
TEMP_nc.sensor_manufacturer_uri = sensor_manufacturer_uri # Sea-Bird
TEMP_nc.sensor_manufacturer_urn = sensor_manufacturer_urn
TEMP_nc.sensor_serial_number = sensor_serial
TEMP_nc.sensor_mount = sensor_mount
TEMP_nc.sensor_orientation = sensor_orientation
4. Écrire les "Global attributes"
2 façons de faire :
- a) apres creation du dataset on instancie les attributs directement "en dur"
fileNC=nc4.Dataset(OutputDirNC+nomficNC,"w", format="NETCDF4", encoding= 'latin-1')
fileNC.description = "CTD profile (NetCDF files) for station "+station_name
fileNC.title = "CTD profile (NetCDF files) station "+station_name+" - Service d’Observation Littoral"
fileNC.keywords = "Seabird CTD temperature, conductivity, fluorimetry, salinity, density, oxygen "
fileNC.history = "Created " + today.strftime("%d/%m/%y")
fileNC.production = "UMR 7999 CNRS / OSU Pytheas UMS3470 CNRS"
fileNC.contact = "Christian Gronz (christian.gronz@labo.fr)"
fileNC.summary = "Mesures CTD : temperature, salinity, oxygen, fluorescence et turbidity 2 profils 0-50 m par jour "
fileNC.featureType = "profile"
fileNC.cdm_profile_variables = "depth,latitude,longitude" ;
fileNC.Conventions = 'CF-1.6, ACDD-1.3'
fileNC.creator_name = 'MIO Marine Institute of Oceanography UMR 7294 CNRS'
fileNC.creator_email = 'maurice.libes@osupytheas.fr'
fileNC.creator_url = 'http://www.osupytheas.fr'
fileNC.creator_type = 'institution'
fileNC.creator_institution = 'OSU Pytheas UMS 3470 CNRS'
fileNC.contributor_role = 'data formatting in netCDF'
fileNC.contributor_url = 'http://www.osupytheas.fr'
fileNC.license = " data are accessible freely and free of cost, for public research and teaching applications...etc."
fileNC.contributor_institution = 'MIO UMR 7294 CNRS / OSU Pytheas UMS 3470 CNRS'
fileNC.institution = 'MIO UMR7294 CNRS / OSU Pytheas'
fileNC.project = 'SOMLIT'
fileNC.publisher_name = 'MIO Marine Institute of Oceanography - OSU Pytheas'
fileNC.publisher_email = 'maurice.libes@osupytheas.fr'
fileNC.publisher_url = 'http://www.osupytheas.fr'
fileNC.publisher_institution = 'MIO UMR 7294 CNRS / OSU Pytheas UMS 3470 CNRS'
fileNC.program = 'SOMLIT Service d’Observation en Milieu Littoral'
- b) utiliser la fonction ncFile.setncattr(attribut, value)
la fonction setncattr() https://unidata.github.io/netcdf4-python/#netCDF4.Dataset.setncattr permet d'instancier un attribut global dans le fichier.
De fait on peut lire des couples "attributs=valeur" dans un fichier CSV externe et les affecter dans le fichier netCDF créé avec la fonction setncattr(attribut, valeur)
- Exemple de fonction pour ecrire les metadonnees globales
def Write_Metadata_NC(ncFile):
#on lit les couples attributs=valeur dans un fichier CSV externe
global_att = pd.read_csv(metadataFileCSV, delimiter='=', header=None, index_col=0)
indexlist = global_att.index
# Creation des global attributs/metadata du fichier NC lues dans un fichier CSV externe
for attribut in indexlist:
value = global_att.loc[attribut].values[0]
value=value.strip()
attribut=attribut.strip()
if attribut == 'Conventions': # le libelle Convention doit avoir un "C" majuscule
ncFile.setncattr(attribut, value)
else:
ncFile.setncattr(attribut.casefold(), value)
ncFile.setncattr('title', 'EMSO Western Ligurian : Bathybot, Microcat sensor (NetCDF files from '+date_deploiement+' )')
ncFile.setncattr("date_created", strtoday)
cprint('Global Attributs OK', color='green', attrs=['bold'])
return 1
exemple de fichier CSV contenant les "global attributes"
date_created = 2023-12-18
Conventions = OceanSITES v1.4,SeaDataNet_1.0,COARDS, CF-1.6
institution_edmo_code =3078
institution_edmo_uri = https://edmo.seadatanet.org/report/3078
institution_ror_uri = https://ror.org/05258q350
geospatial_lat_max = 42.8015
geospatial_lat_min = 42.8014
geospatial_lon_max = 5.9828
geospatial_lon_min = 5.9826
geospatial_vertical_max = 2420
geospatial_vertical_min = 2420
time_coverage_start = 2023-04-28T00:00:00Z
time_coverage_end = now
update_interval = daily 24 hours
site_code = Bathybot
emso_facility = Western Ligurian Sea
source = sea bed vehicle
platform_code = ""
wmo_platform_code = unknown
format_version =1.4
network = EMSO-ERIC
4.1 la convention CF et les global attribute
Note
Attention la convention CF a quelques exigences sur les métadonnées présentes dans un fichier netCDF !
- FeatureType
Il faut obligatoire instancier un attribut featureType
qui indique le type d'échantillonnage des données (point, TimeSeries, Profile, Trajectory, etc...)
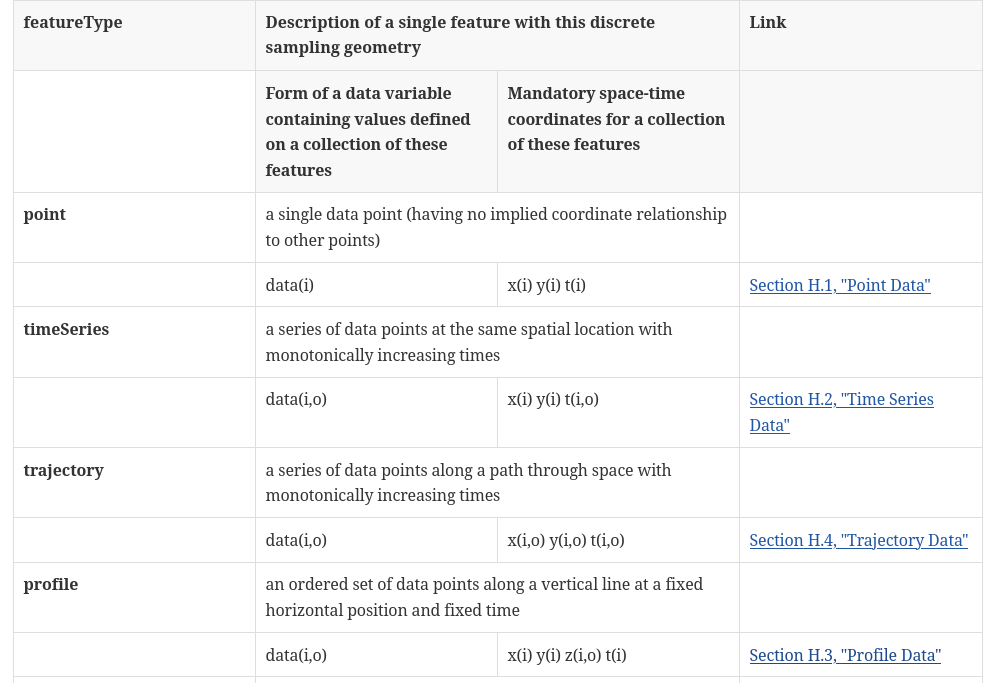
- cf_role
Dans la mesure du possible, il convient d'inclure une variable de coordonnées ou de coordonnées auxiliaires avec l'attribut cf_role.
Les seules valeurs acceptables de cf_role pour les ensembles de données CF de géométrie discrète sont timeseries_id , profile_id et trajectory_id . La variable portant l'attribut cf_role peut avoir n'importe quel type de données. Lorsque cet attribut est attribué à une variable, celle-ci doit fournir un identifiant unique pour chaque instance d'élément.
-
Les fichiers CF qui contiennent des types de caractéristiques timeSeries, profile ou trajectory ne doivent comporter qu'une seule occurrence d'un attribut cf_role ;
-
les fichiers CF qui contiennent timeSeriesProfile ou trajectoryProfile peuvent contenir deux occurrences, correspondant aux deux niveaux de structure de ces types de caractéristiques.
5. Ecrire les données dans la section "data"
 : écrire les données dans chaque variables netcdf créée, revient à affecter/transférer un tableau de données DANS la variable netCDF précédemment créée
: écrire les données dans chaque variables netcdf créée, revient à affecter/transférer un tableau de données DANS la variable netCDF précédemment créée
Syntaxe: variable_netcdf[:] = tableau_donnees_prog_python
# cas d'une variable chaine de caractere : il faut convertir la chaine en tableau
nomstation=”JULIO” #string
stationname[:] = nc4.stringtoarr(nomstation,len(nomstation))
# cas de la variable "time", il faut convertir une date chaine de caractere en format numerique
dateprofile=”2017-07-12T10:30:01”
NC_TIME_FMT = 'seconds since 1970-01-01T00:00:00Z'
time[:] = nc4.date2num(dateprofile, units=NC_TIME_FMT)
# on affecte la variable scalaire
lats[:]=latitude
lons[:]=longitude
Pour écrire les données dans les variables netcdf, on transfere un tableau de valeur dans la varaible netcdf créée
# variable_netcdf = vecteur/tableau de données
tab_temp=[37.2,37.3,38.1,36.4...]
temp[:] = tab_temp
conductivity[:] = tab_conductivity
salinity[:] = tab_salinity
irradiance[:] = tab_irradiance
fluorescence[:] = tab_fluorescence
fileNC.close()
TP programme python - API
On va faire un programme qui lit un fichier de données au format CSV, et le transforme en fichier netCDF
On va dérouler les différentes parties d'un programme simple avec l'interpéteur bpython:
a) créons le dataset vierge à partir d'un fichier CSV
-
placez vous dans le repertoire ~/tp
- cd ./tp -
Lancez l'interpréteur
bpython
bpython version 0.22.1 on top of Python 3.10.12 /usr/bin/python3
>>>
# importation des librairies necessaires
import netCDF4 as nc4
import pandas as pd
import numpy as np
from datetime import datetime
import os
# ouverture du fichier CSV avec pandas
# on cree une matrice pandas "df" (dataframe)
fileName="Giens_2023-01.csv"
df = pd.read_csv(fileName,skiprows=3)
print(df)
print(df.columns)
# conversion des dates et tri du fichier si besoin
df['UTCDateTime']=pd.to_datetime(df['UTCDateTime'],yearfirst=True)
df.sort_values(by="UTCDateTime", axis=0, ascending=True,inplace=True, na_position='first')
# creation du fichier nc vierge
base,ext=os.path.basename(fileName).split('.')
netCDFFileName=base+'.nc'
ncfile = nc4.Dataset(netCDFFileName,'w', format='NETCDF4')
b) créons les "global attributes
Pour rappel les "global attributes" sont les métadonnées générales qui décrivent les données présentes dans le fichier.
La maniere la plus "directe" pour créer ces métadonnées
est de les instancier directement dans le code.
Si ncfile est le descripteur du fichier netCDF qu'on vient de créer précédemment, alors
ncfile.nom_attribut = valeur_attribut
créera l'attribut "nom_attribut'
# Exemple : Direct METADATA in code
today=datetime.now()
nom_station="Giens"
ncfile.created = "Created file : " + today.strftime("%d/%m/%y")
ncfile.history = "fichier produit par programme python"
ncfile.title = "HTMNET Data for station "+nom_station
ncfile.summary = "blabla résumé"
ncfile.doi = "https://doi.org/xxxx/yyy"
ncfile.licence = "CC-BY-04"
etc. etc.
lorsqu'on a beaucoup de métadonnées globales, le fait de les insérer dans un fichier séparé et de les intégrer par programme python, rend le programme plus modulaire et simple a entretenir.
Dans ce cas on lit des couples "attributs=valeur" dans un fichier CSV externe Global-Attributes.csv
## attribut = valeur
Instrument_Microcat=SDN:L22::TOOL1457 http://vocab.nerc.ac.uk/collection/L22/current/TOOL1457
institution = MIO UMR 7294 CNRS / OSU Pytheas
contributor_name = maurice.libes@osupytheas.fr
keywords = Biogeochemistry,GeoSciences, oxygen, optode, temperature, pressure, salinity, fluorescence
etc etc.
et on les affecte dans le fichier netCDF créé avec la fonction setncattr(attribut, valeur)
#exemple pour un attribut... faire une boucle si on les lits dans un fichier
attribut="institution"
valeur = "MIO UMR 7294 CNRS / OSU Pytheas"
ncfile.setncattr(attribut, valeur)
c) créons les dimensions
c'est une serie temporelle, on met la variable "time" comme dimension (variable time qu'on creera juste apres) on met aussi la longueur de la chaine de caracteres en dimension
ncfile.createDimension('time', None)
# pour les chaines de caractere il faut definir la dimension de la chaine
nom_station="Giens"
lenstation=len(nom_station)
ncfile.createDimension('station_name_len', lenstation)
d) créons les variables
# variable time
time = ncfile.createVariable('time', 'i4', 'time')
NC_TIME_FMT = 'minutes since 2000-01-01 00:00:00 UTC'
time.units = NC_TIME_FMT
time.standard_name = "time"
time.units = NC_TIME_FMT
time.calendar = "standard"
time.CoordinateAxisType = "Time"
time.axis = "T"
time.coverage_content_type = "coordinate"
# variable "station" en chaine de caracteres
station_name = ncfile.createVariable('station_name', 'S1', 'station_name_len')
station_name.long_name="nom de la station"
# le cf_role est demandé par la convention CF
station_name.cf_role = "timeseries_id"
# variable temperatur de l'eau
tmer = ncfile.createVariable('tmer', 'f4', 'time')
tmer.standard_name = "sea_surface_temperature" ;
tmer.long_name = "SST"
tmer.units = "degree_C"
e) affectons les valeurs des variables netcdf
pour mettre les valeurs dans une varaible netcdf... il faut constituer un tableau de valeurs dans le programme, qu'on affecte en une seule opération à la variable netcdf
variable_netcdf[:]=tableau_de_valeurs
# exemple pour la variable time tableau 1D
pour la variable time il faut convertir les dates "chaines de caracteres" en un format numérique avec la fonction `date2num`
timeVector=[]
for ind in df.index:
timeValue=nc4.date2num(df['UTCDateTime'][ind],units=NC_TIME_FMT,has_year_zero=False,calendar='standard')
timeVector.append(timeValue)
# on affecte le tableau dans la variable nc
time[:]=timeVector
# pour la variable station chaine de caracteres
il faut convertir la chaine de caracteres en un tableau de caracteres avec la fonction `stringtoarr`
station_name[:]=nc4.stringtoarr(nom_station,len(nom_station))
# pour la variable tmer on affecte la colonne "wT" du dataframe pandas
tmer[:]=df['wT']
# on ferme le fichier pour dumper sur disque
ncfile.close()
- verifiez votre fichier netcdf créé avec panoply ou ncview
panoply Giens_2023-01.nc
Le programme complet genereNetCDF.py est disponible dans le repertoire ~/tp/python-netcdf
(NB: pour minimiser le code python, nous n'avons volontairement pas tenu compte de la standardisation pour produire du netcdf interopérable)
- exemple programme python de conversion netcdf : programme python genereNetCDF.py
- Usage :
- $ genereNetCDF.py -n Giens_2023-01.csv
-
exemple de fichier Metadata externe : fichier Metadata
-
exemple fichier de données CSV : fichier data CSV
- le fichier netCDF produit : fichier netcdf
$ ncdump Giens_2023-01.nc | more
ou
$ panoply Giens_2023-01.nc
Liens utiles
-
Documentation de l’ API Python interface à la librairie netCDF
-
Les attributs netCDF